转录组数据分析01-比对
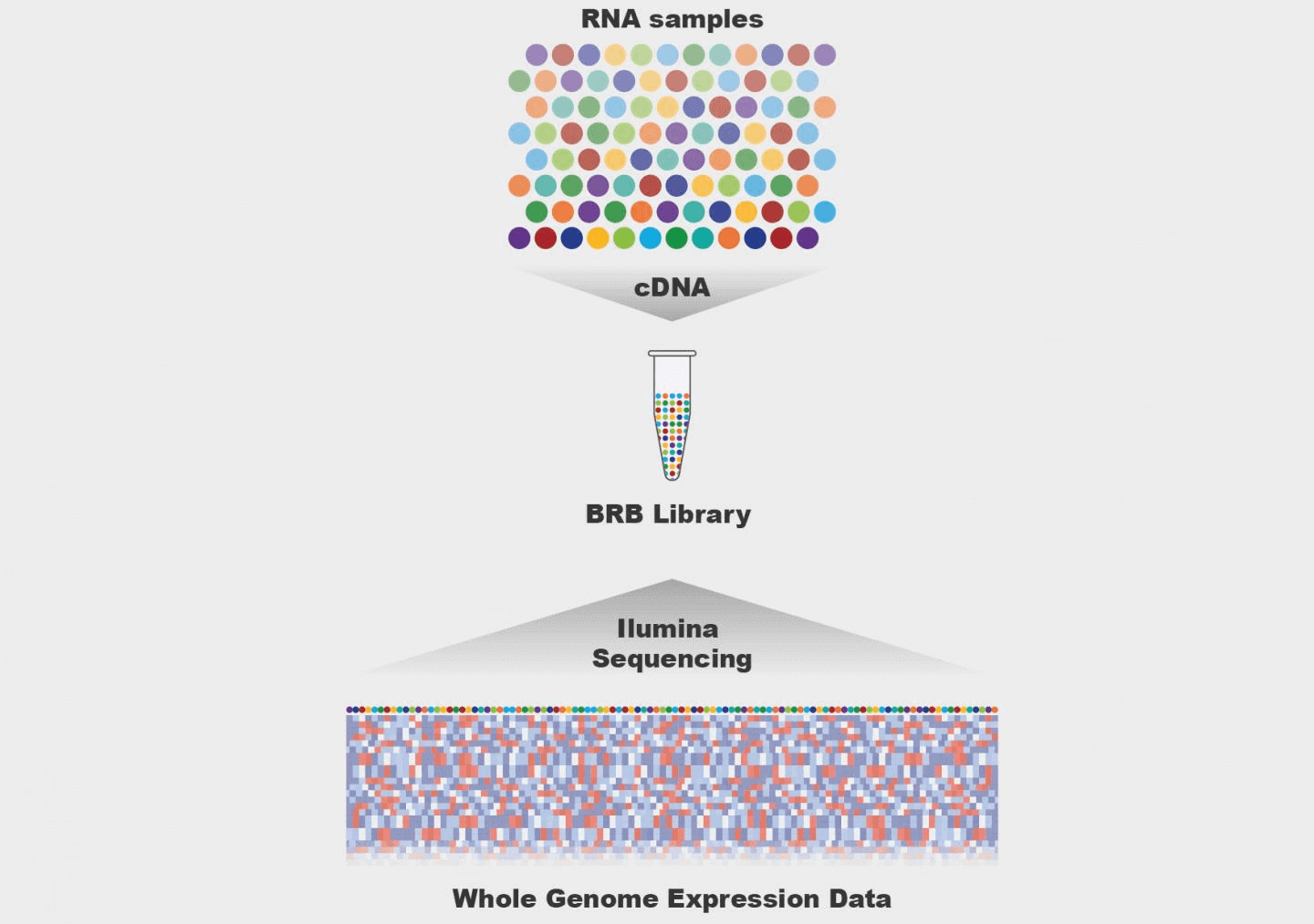
最近有一批转录组数据需要分析,从公司拿到raw data之后进行后续分析。目前转录组测序十分成熟,测序质量都很高,所以我这里就简单地质控一下,由
fastp完成。
gganimate:数据跳动
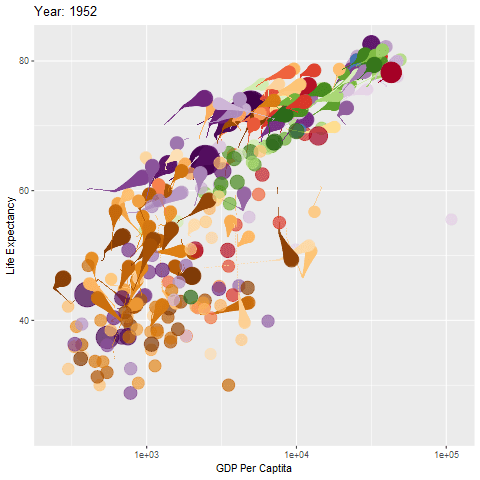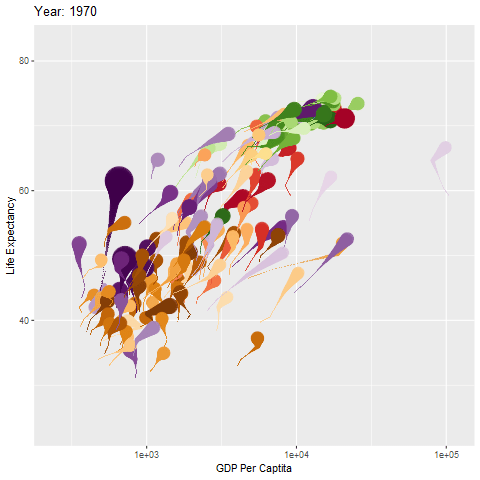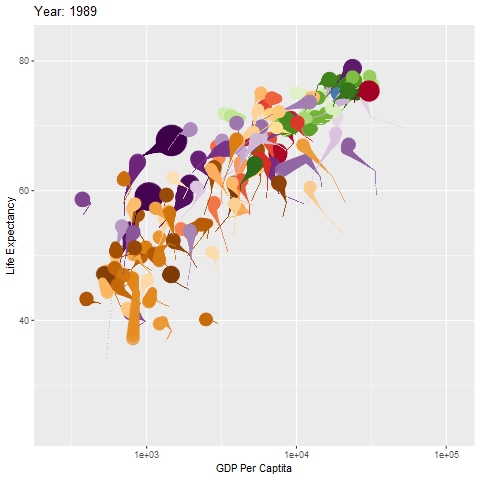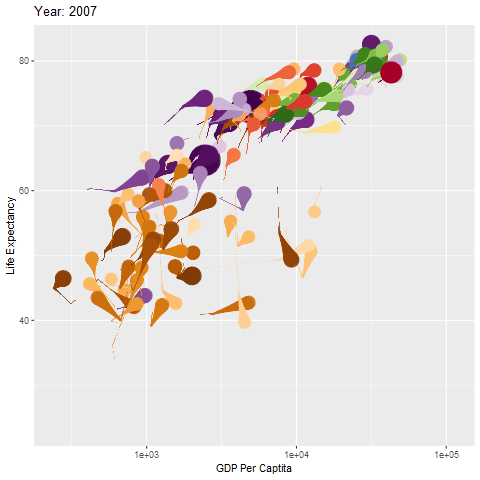
简介
前面已经粗略学了一下gganimate的部分语法,下面继续。
gganimate:让数据动起来

简介
一年多以前我就写了篇R语言动画的博客:
R语言可视化学习笔记之gganimate包。当时使用的是由
David Robinson开发的gganimate。后面由
Thomas Lin Pedersen接手,推倒重来,拓展了很多动画语法。具体的可参考
Thomas Lin Pedersen的
gganimate的Github主页。
rehh:全基因组选择印记扫描

简介
Sabeti在2002年的时候提出了一种叫做Extended Haplotype Homozygosity (EHH)的统计方法,用来扫描全基因组范围内的选择印记。在此基础上又衍生出了iHS、XP-EHH以及Rsb等统计方法。R包rehh实现了这几种统计方法的封装。
Genome Biology | 大豆、玉米驯化过程中AT碱基含量显著上升
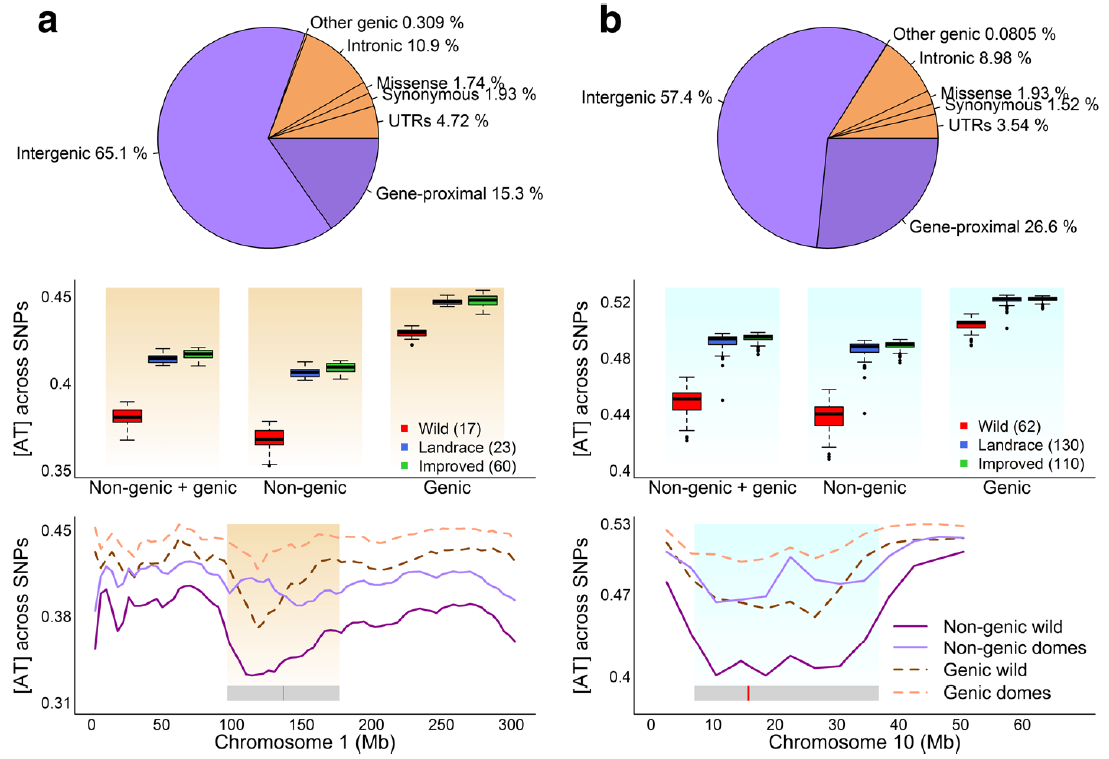
一句话总结
大豆、玉米驯化过程中AT碱基含量显著上升。
利用EMMAX进行GWAS分析
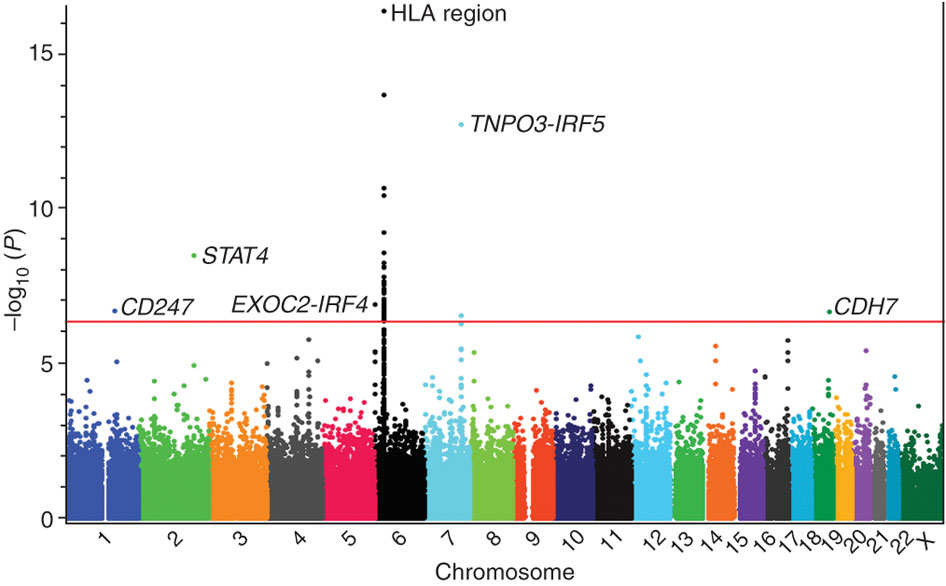
文件准备
利用EMMAX进行GWAS分析需要以下文件
Circos配置文件详解

Circos的配置文件准备
Circos的使用主要通过输入一个配置文件,改配置文件的主要内容以各种区块表示,大区块中可以包含小区快。区块中以"变量=值"的方式来进行参数的设定:
<links>
<link>
file = data/set1.txt
color = black
...
</link>
<link>
file = data/set2.txt
color = red
...
</link>
</links>
有些配置文件不需要改动,比如颜色、字体等,一般将这类信息保存到一个独立的配置文件中。只需要在主配置文件中声明包含这些独立的配置文件名及其路径,即表示使用这些配置信息。最常用的放置到主配置文件尾部的数行
三种方法在地图上绘制网络图

最近为了绘制几幅简单地图,查阅了一些资料,看到了Markus konrad的 帖子,非常赞。其中他的部分思路对于我们学习可视化很有帮助。

