NP|油菜泛基因组及生态型分化

1. 简介
油菜(Brassica napus)是世界上重要的油料作物,起源于地中海地区,大约是在7500年前由白菜(Brassica rapa)和甘蓝(Brassica oleracea)自然杂交加倍形成的异源四倍体。与白菜以及甘蓝的大量杂交,极大地扩宽了油菜的遗传多样性。为了适应自然季节变化比如春化需求、耐冬性以及光响应开花,油菜形成了不同的生态型:冬性油菜(WORs)、半冬性油菜(SWORs)以及春性油菜(SORs)。目前油菜已经公布了4个版本的参考基因组(冬油菜:Darmor-bzh,Tapidor以及半冬油菜:ZS11,NY7)。但是由于测序方法或者测序深度的限制,已公布的基因组的准确度以及完整度都不足以支撑结构变异(SV)的研究,而结构变异往往决定了遗传多样性以及重要的农艺性状。泛基因组(pan-genome)指的是一个物种所有基因的集合,包括核心基因集(core genes)以及非必需基因集(dispensable genes)。多个高质量的参考基因组有利于油菜基因组结构研究以及探究油菜不同生态型分化的遗传基础。
数据库安全漏洞修复

由于
实验室网站需要开通外网服务,学校安排了安全漏洞检测,共发现安全漏洞3个,其中高危0个、中危1个、低危2个。存在的安全隐患主要包括目录浏览、X-Frame-Options Header未配置等安全漏洞,可能将造成用户信息泄露等危害。
构建Consensus sequence
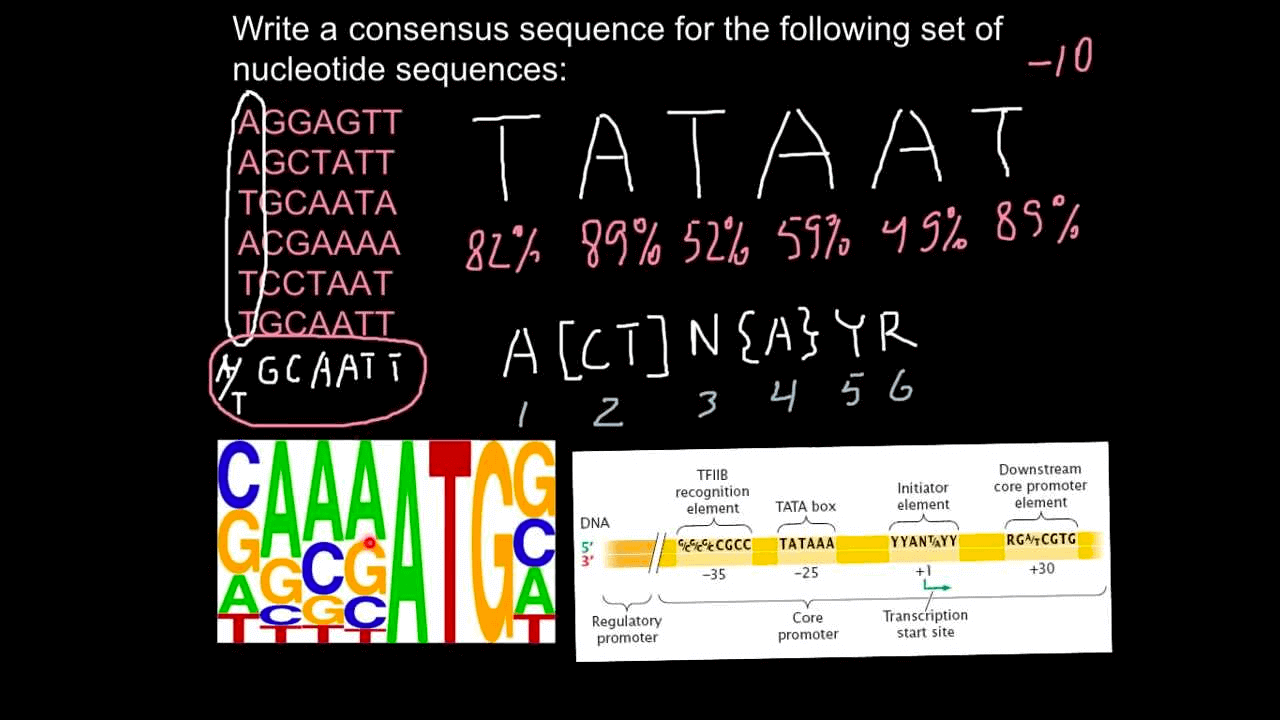
简介
Consensus sequence又称为一致性序列、保守序列、共有序列等。维基百科的解释:
In molecular biology and bioinformatics, the consensus sequence (or canonical sequence) is the calculated order of most frequent residues, either nucleotide or amino acid, found at each position in a sequence alignment.
EDTA:转座子注释
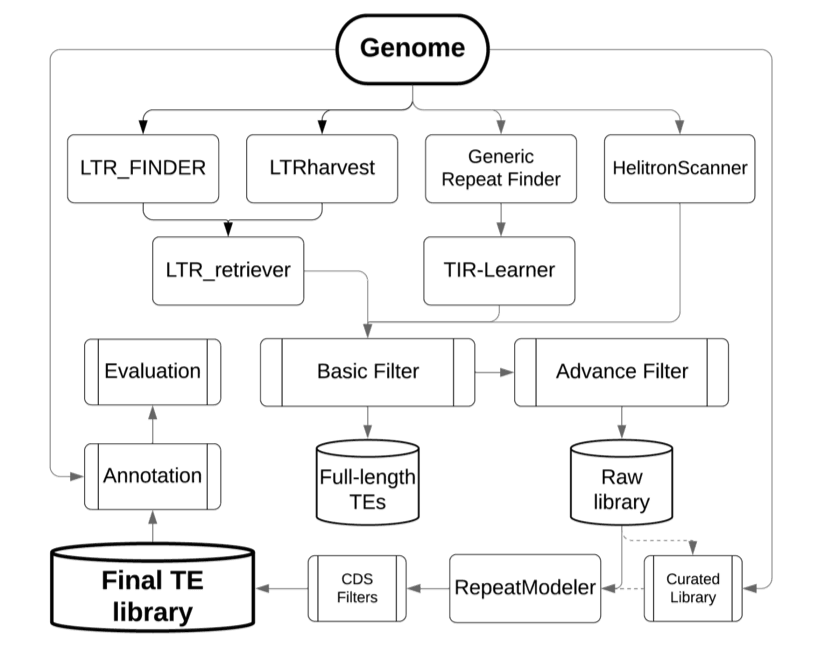
简介
转座子注释目前有非常多的软件,而EDTA(The Extensive de novo TE Annotator)整合了大部分目前常用的转座子注释软件,值得注意的是EDTA是全基因组从头注释。EDTA最大的好处就是简单实用,而且经过大量的改进,目前在安装、运行以及结果解释等方面十分完善。
Shell | 多线程并行计算

多线程并行计算可以提高效率,节省时间。最近工作中有一批数据需要处理,学习了一下批量多线程操作。
Nature Reviews Genetics | 测序时代的基因组转座子
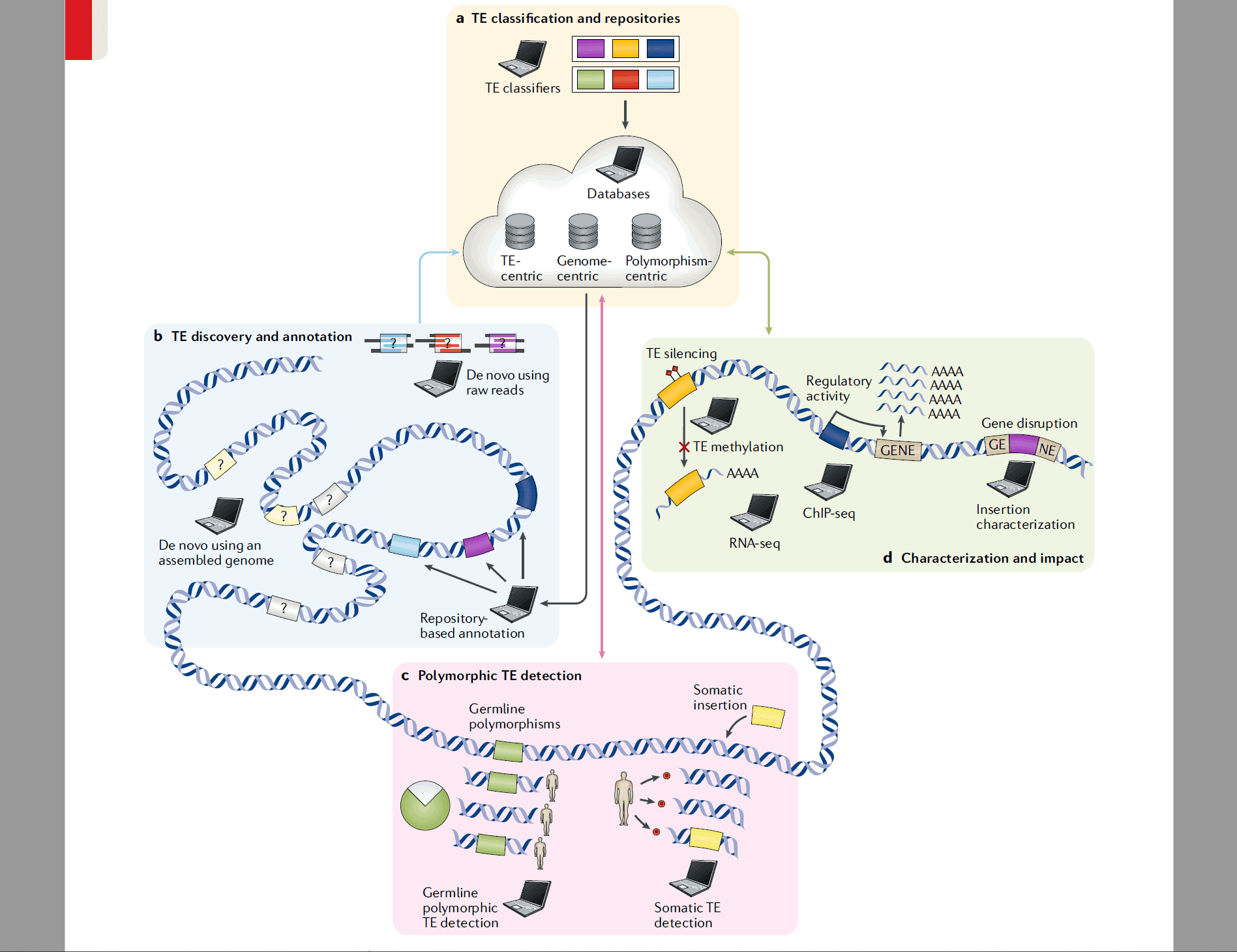
简介
许多物种的基因组大部分是来自于转座子(Transposable elements, TEs)。此外,通过各种自我复制机制,TEs在大多数物种基因组中持续增殖。TEs会影响调控、转录以及新蛋白形成进而与性状比如疾病相关联。尽管TE表现出了显著的影响,但是很多基因组研究还是将它剔除在外,这主要是由于它具有高度的序列重复性,导致研究变得十分复杂。幸运的是随着大量的方法以及计算机软件的开发,TE的研究逐渐深入。本综述总结了TE研究的相关计算工具并着重指出了未来的TE研究存在的挑战与空白。对TE进行基因组上全面的分析不仅仅是“掩盖”这些重复序列(This Review presents a summary of computational resources for TEs and highlights some of the challenges and remaining gaps to perform comprehensive genomic analyses that do not simply “mask” repeats)。
SpeedSeq:一个快速基因组分析和注释的灵活框架
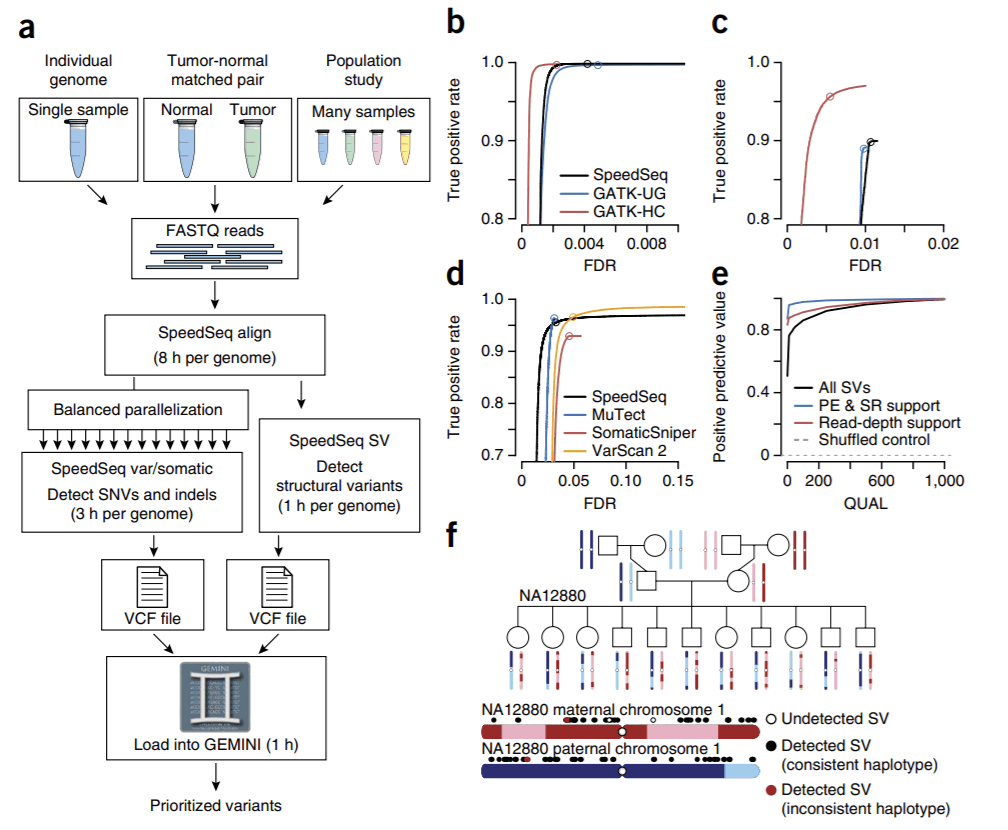
简介
Speedseq是一个快速基因组分析和注释的灵活框架,发表在 Nature Method上封装了大量基因组分析的软件,比如比对软件BWA,calling SNP软件freebays,SV鉴定软件lumpy等,这也导致了这款软件的安装十分繁琐。另外SpeedSeq基于Python2环境,目前很多新版本的模块比如SpeedSeq依赖的numpy、pysam以及scipy基本不支持Python2了,所以如果你基于 SpeedSeq主页的安装手册进行安装各种依赖,会让你怀疑人生的。此外以后如果有软件是依赖Python2环境的,我的建议是尽量别用了,除非是找不到替代的软件了。
Nature Reviews Genetics | 测序时代的基因组结构变异
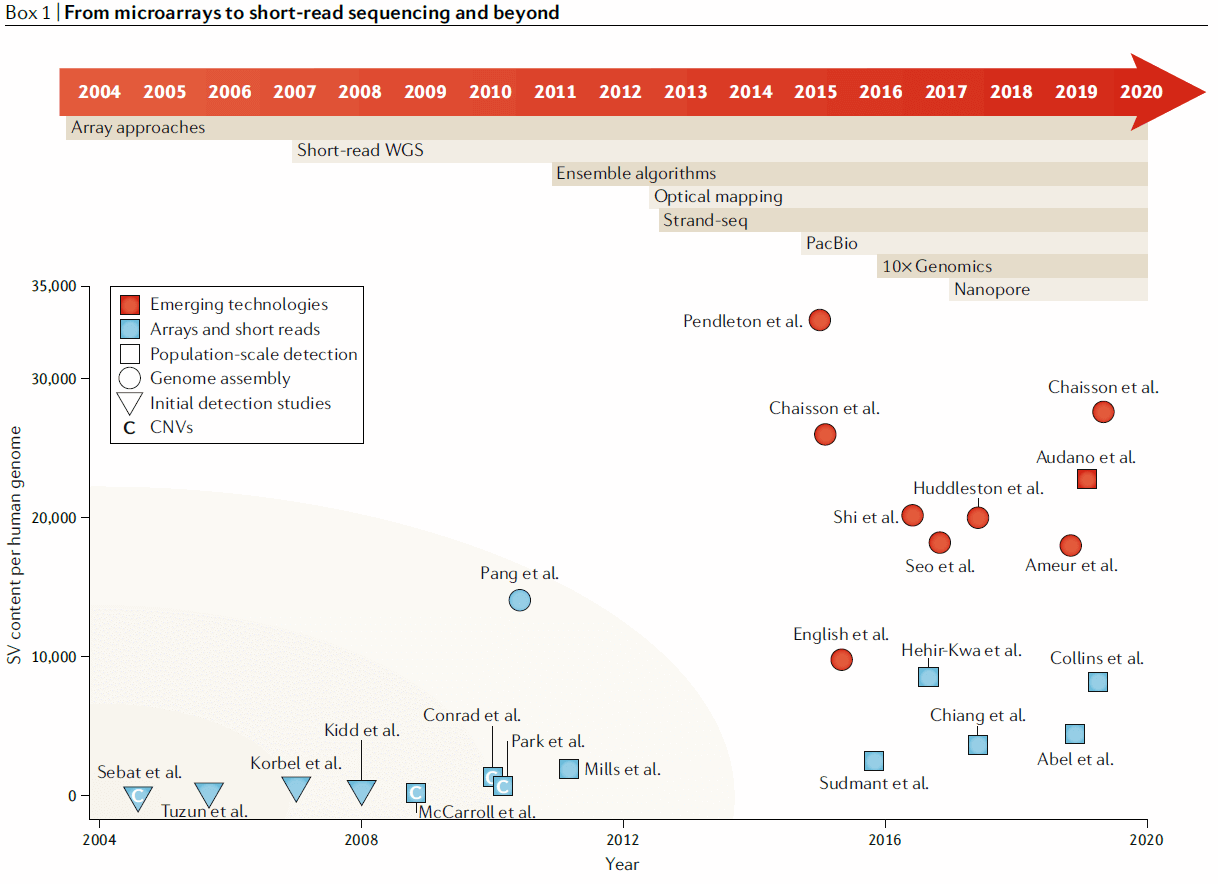
简介
结构变异(SV)的鉴定对于解析基因组是十分重要的。得益于检测算法以及测序技术的发展,全基因组结构变异分析成为可能。本文作者综述了基因组结构变异的检测方法,同时指明了整合基因组结构变异以及生物学信息的研究方向。
