Genome Biology | 大豆、玉米驯化过程中AT碱基含量显著上升
一句话总结
大豆、玉米驯化过程中AT碱基含量显著上升。
背景
作物驯化大都集中在基因、遗传多样性、基因组结构以及表观基因组等。本文比较有意思的是从全基因组AT碱基含量角度解释作物驯化。
结果
作者首先从已发表的文章中获取了100份玉米群体(包括17份野生种、23份地方品种以及60份改良品种)、302份大豆群体(包括62份野生种、130份地方品种以及110份改良品种)的SNP。并在此基础上划分成两份SNP数据集(common SNP set and population-private SNP set)。
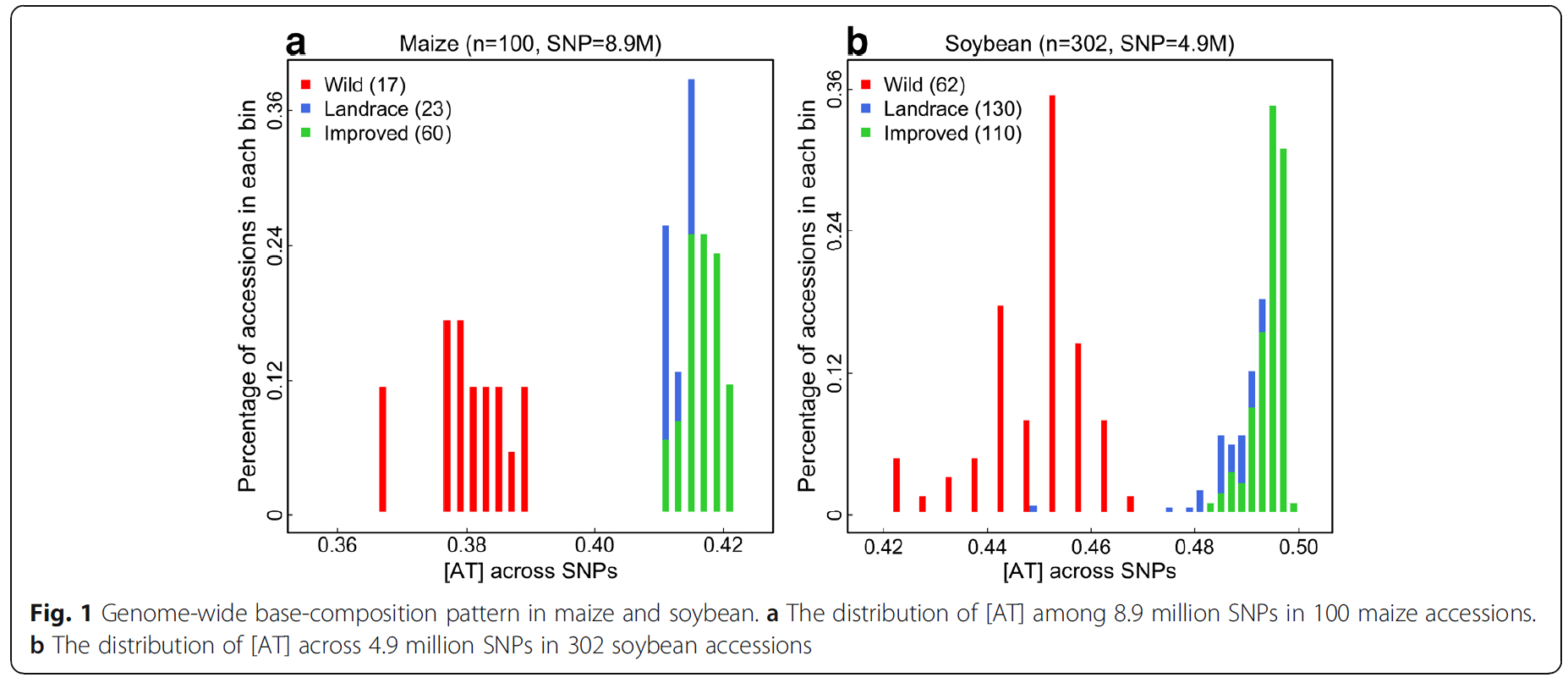
全基因组水平上AT增加
在全基因组common SNP set也就是所有品种都含有的SNP中,根据统计每个品种的AT含量,基于single-strand parity rule 2 (PR2)密码子偏好性([A] ≈ [T] and [G] ≈ [C]),发现大豆和玉米的驯化品种中AT含量都显著升高。
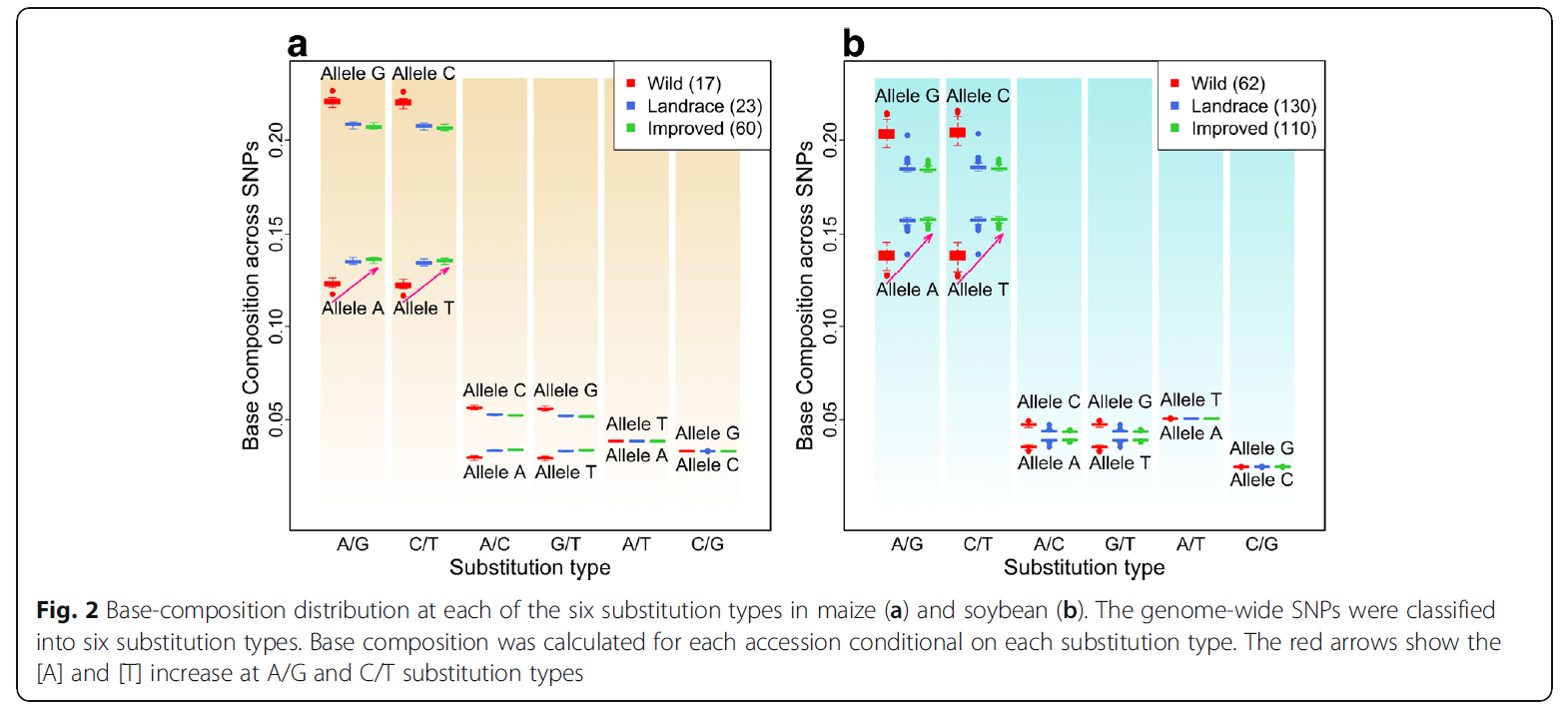
DNA碱基替换类型中的碱基组成
碱基替换有6种类型:两种转换(A/G and C/T),四种颠换(A/C, A/T, C/G, and G/T),发现6种碱基替换对AT含量升高的贡献是不一样的,得益于高发生频率,两种转换(A/G and C/T)是主要的贡献者。
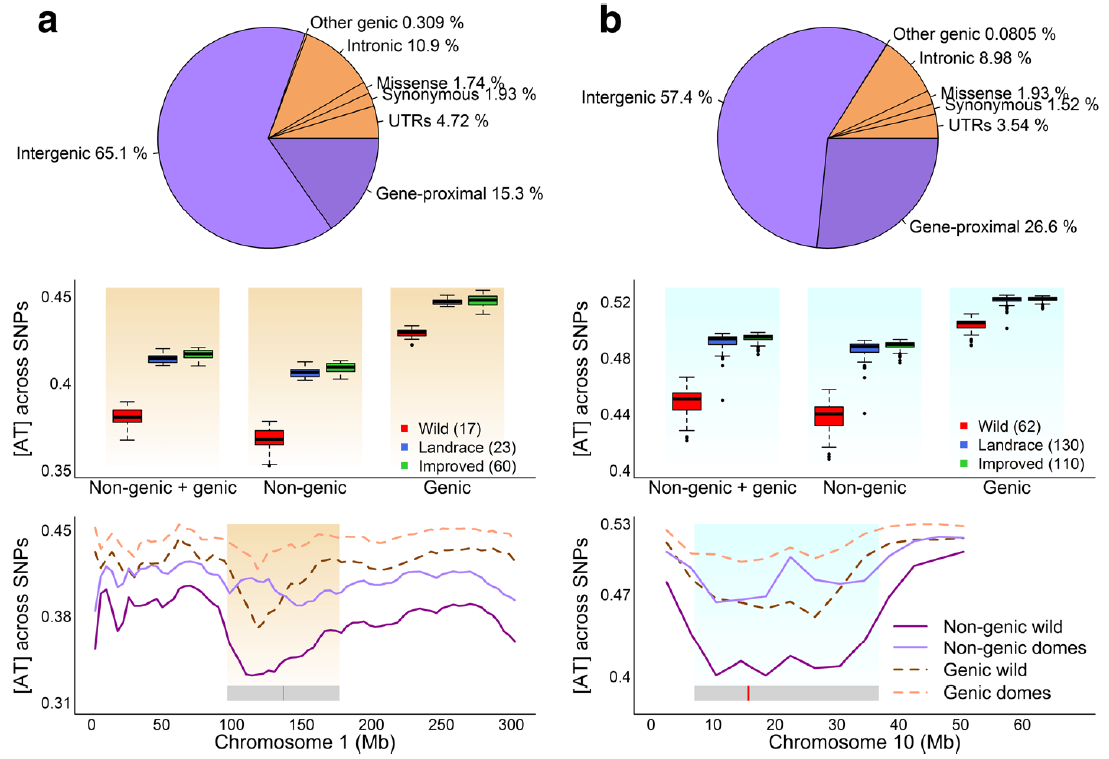
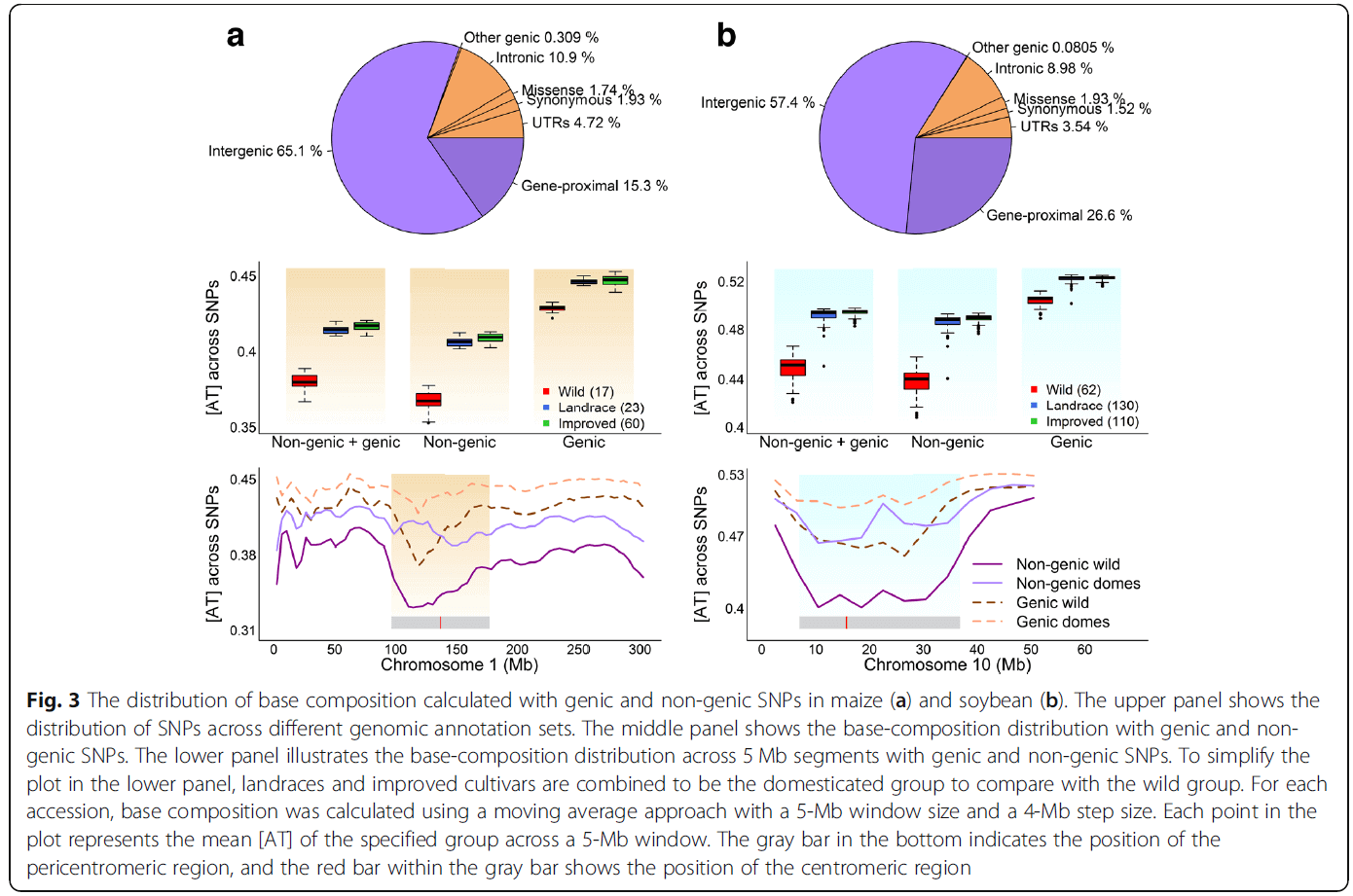
基因组不同区域碱基组成模式
统计每个品种基因区(genic)以及非基因区(non-genic)AT含量,发现驯化品种相比野生品种,基因区以及非基因区AT都明显更高,非基因区对AT含量增加的贡献更大,这可能是由于非基因区SNP远远多于(4~5.5倍)基因区SNP,所以作者又随机抽取基因区以及非基因区等量SNP进行统计,发现结果一致。另外还发现基因区含有更多高MAF的SNP,说明相对于非基因区,基因区相对保守。我们知道着丝粒周围基因密度相对较低,所以作者又统计了着丝粒附近的AT含量,发现着丝粒附件AT含量急速扩增,猜测可能是由于着丝粒区域含有大量的重复序列、转座子以及更低的重组率等。接下来作者又对选择性清除区域进行统计发现野生种与驯化种在选择性清除区域AT含量差异远远大于非选择性清除区域。说明驯化进程对多态性位点碱基组成是有影响的。
solar-UV信号相关的motif富集
为了检测SNP的产生在不同的序列是否有偏好性,作者根据SNP以及其上下游各一个碱基组成的motif进行分析。根据6种碱基替换以及4种碱基,作者将SNP划分为96个3碱基motif。按照概率,96个motif产生的概率应该是1/96。在玉米以及大豆中作者共检测到了14个motif的概率高于1/96,其中11个是属于A/G以及C/T转换类型的。同时发现solar-UV相关信号的motif是存在着碱基偏好性的。
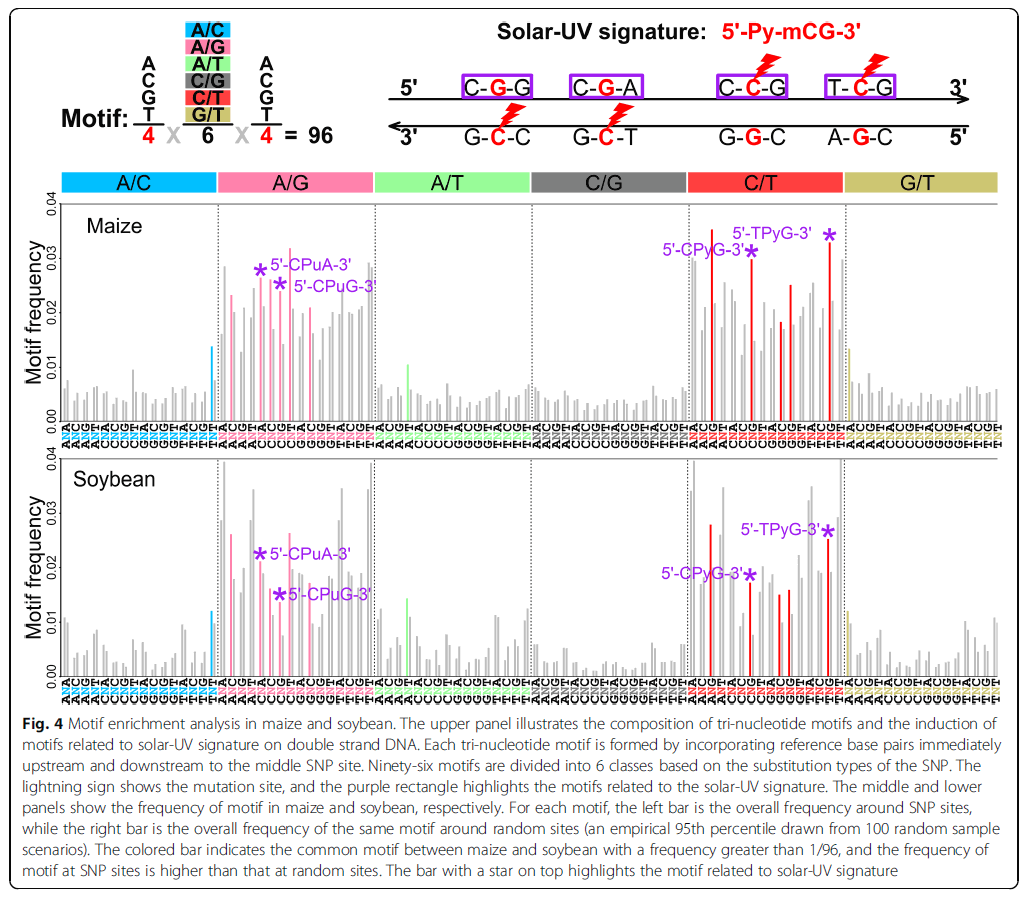
群体特异SNP中的突变普
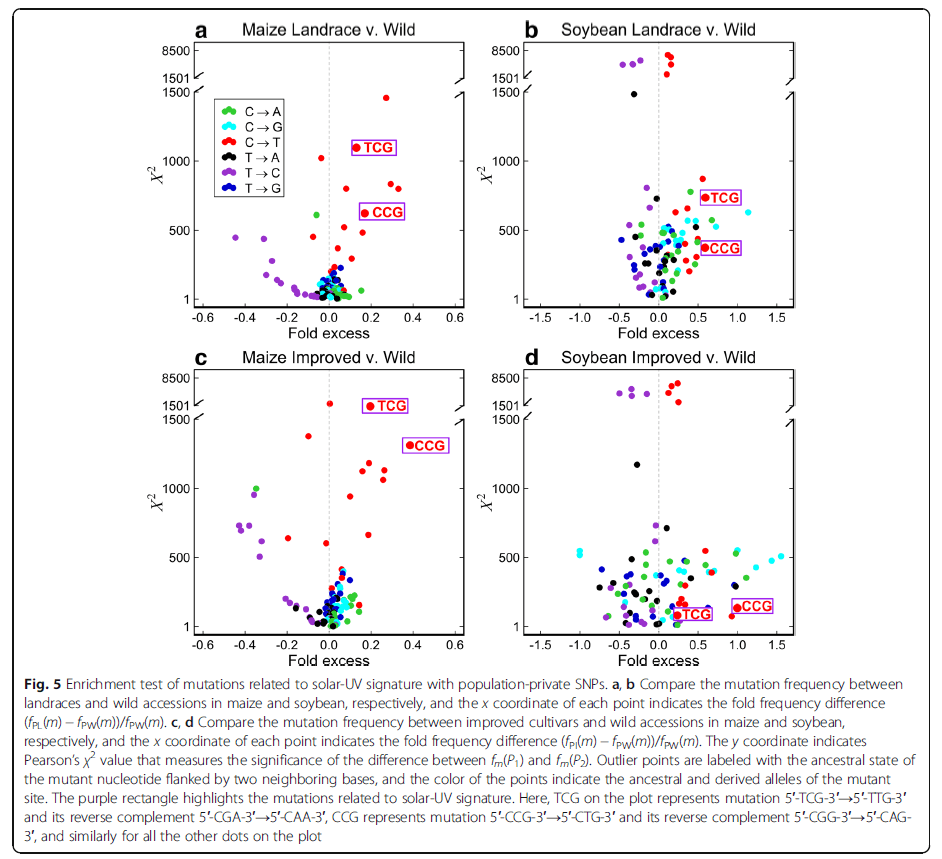
接下来作者又对群体特异SNP(population-private SNPs)进行分析以验证是不是跟solar-UV信号相关的motif是集中在最近发生的突变中。至于如何区分出population-private SNPs,原文说得十分清楚
These private SNP sets are different from the earlier common SNP sets with a small overlap. A SNP is considered as population private if it is segregating in 1 lineage but fixed ancestral allele in other lineages.
对于每一种作物,作者获取了四类群体特异性SNP数据集:
- 野生种特异的SNPs(PW)
- 驯化种特异的SNPs(PD)
- 地方品种特异的SNPs(PL)
- 改良品种特异的SNPs(PI)
PW表示在野生种中分离但是在地方品种以及改良品种中固定的SNPs,其它以此类推,分析这些SNPs数据集有利于评定这些家系在最近的共同祖先分化之后的突变率。
基于前面分析的96种motif,作者继续在群体特异性的SNP中进行分析,发现相比于野生种,与solar-UV信号相关的5′-TCG-3′→5′-TTG-3′以及5′-CCG-3′→5′-CTG-3′在驯化种中具有更高的突变率,同时对基因组不同区域进行群体特异性的SNP分析出来的结果与前文一致。
在群体特异性的SNP以及common SNP中发现solar-UV信号相关的突变比例过高说明驯化过程中solar-UV可能是导致AT含量增加的一个主要因素。
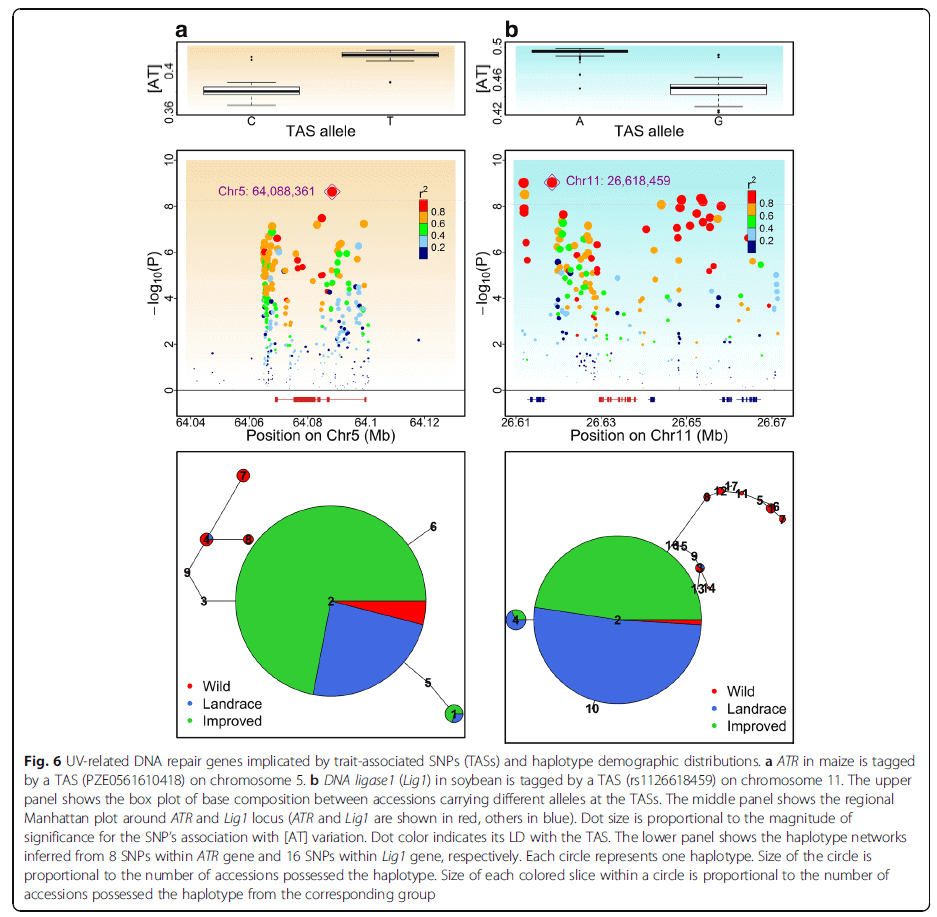
AT含量增加的基因主要为UV-damaged DNA修复基因,这些基因与基因组分化有关
人类的GWAS研究发现DNA修复相关基因与基因组的分化有关,因此作者猜测UV-damaged 修复通路上的基因附近的位点是不是与基因组上的AT分化有关,为此,作者将从common SNP中获取的AT值作为表型进行GWAS,鉴定到了大量UV-damaged DNA修复相关的基因。接下来作者挑选了几个重要基因进行深入分析,分析了AT含量变化、单体型以及网络分析,以此来说明AT含量的变化与基因组的分化有关。
这篇文章立意新奇,不从基因、遗传多样性、基因组结构以及表观基因组方面入手,反而从基因组AT含量切入,有理有据,条例清晰地阐明了大豆、玉米驯化过程中AT碱基含量显著上升这一结果。具体可参考 原文。